本栏目由医隆网专家团队审核!
基因组包含了整个生物体的全部遗传信息,因此,获得生物体基因组的全部序列对于进行生命科学研究具有十分重要的现实意义。测序分析能够为基因DNA序列提供最真实可靠的信息,它可以比较全面地描述基因的复杂性和多样性。1953年,剑桥大学卡文迪许实验室Watson和Crick提出DNA双螺旋结构模型以后,人们开始探索研究 DNA一级结构的方法,由此开启了分子生物学时代。因为这一巨大贡献,1962年Watson、Crick和Wilkins共同分享诺贝尔奖。但是由于没有找到分别降解四种脱氧核糖核酸的专一酶,很长时间内都只能通过测定RNA序列来推测DNA序列。所用方法是先将RNA用酸水解或外切酶降解,再经双向电泳同系层析将其分开。这种方法既费时又不准确,测定10~20个核苷酸序列往往要花费1~2年的时间,限制了人类对自身DNA序列的了解。
1953年,英国剑桥大学Frederick Sanger在对牛胰岛素的研究中首先提出氨基酸直接测序概念,他因此而获得1958年诺贝尔化学奖。1977年,他发明双脱氧核苷酸末端终止法,并由于这一技术创新于1980年第二次获得诺贝尔化学奖。此外,美国哈佛大学Walter Gilbert等发明的化学降解法,这些技术发明标志着第一代测序技术的诞生。这两种技术都是根据核苷酸在某一固定的点开始,随机在某一特定碱基处终止,进而产生A、T、C、G四组不同长度的核苷酸,通过PAGE胶电泳获得DNA序列。20世纪80年代中期基于双脱氧终止法的自动测序仪使用非放射性荧光标记代替同位素标记,通过激光激发荧光反应并通过探测器收集荧光信息,通过图像识别和分析自动获得DNA序列。1987年美国Applied Biosystems(ABi)公司推出第一台自动化测序仪ABI 370,产量达到每天1000个碱基。这种自动化技术不断升级直到1997年推出ABI 3700能够使用96道毛细管阵列同时检测96个样本,产量达到每天500 000~1 000 000个碱基。测序技术发展也给人类认识自身基因序列提供了可能。1986年,诺贝尔奖获得者Renato Dulbecco在Science上发表文章《肿瘤研究的转折点:人类基因组测序》,提出了认识人类基因组对于了解疾病的重要性。1987年初,美国能源部和国立卫生研究院为人类基因组计划(Human Genome Project,HGP)下拨了启动经费并在1990年经过美国国会批准正式启动,之后英国、日本、法国、德国、中国相继加入该计划。1999年中国科学院遗传研究所人类基因组中心注册参与国际人类基因组计划,承接了3号染色体1%基因组测序任务。2000年6月26日人类基因组工作草图完成,2003年4月,包含基因序列中的98% (原预计为 95%)获得了测定,精确度为99.99%。
在几十年的发展中,尤其在人类基因组完整图谱绘制完成后,基因组学、转录组学等新兴研究领域相继涌现,它们的研究成果大大加深了人类对生命遗传机制和疾病发生等复杂过程的认识。尽管人类基因组计划使得我们对于疾病发生机制的了解更加深入,但是该项目历时15年,耗费30亿美元。对于基因组和疾病研究检测需要简便快捷和成本低廉的大规模测序方法。虽然Sanger测序技术在测序准确性和读长方面已经相当出众,但其测序通量与速度低、测序成本高等缺点导致它无法满足大规模测序任务的要求,这促使第二代测序技术的诞生。利用了Sanger中的可中断DNA合成反应的dNTP,一些其他测序技术也得到快速发展,如焦磷酸测序法、链接酶法等。1998年Jonathan Rothberg报道了焦磷酸测序技术,这一技术是依赖核苷酸合成时焦磷酸释放,该技术无需电泳检测,操作简单。自2005年以来,454 Life Sciences公司推出了第一种第二代测序技术平台——Genome Sequencer 20 System,这一平台是基于焦磷酸测序法的超高基因组测序系统,其产量可以达到20 000 000个碱基。1998年英国剑桥大学Osborne和Barnes成立Solexa公司,2006年其仪器1G Genome Ananlyzer( GA)正式产出数据,这一测序平台是基于DNA簇、桥式PCR和可逆阻断等核心技术实现边合成边测序,其通量能达到1Gb。2007年,Illumina公司以6亿美元的高价收购了Solexa,使GA得以商品化。2005年,第一代美国Applied Biosystems( ABi)公司收购了进行测序技术开发的公司Agencourt,其技术来源于哈佛大学George Church。2006年RadojeDrmanac等成立Complete Genomics公司,其测序平台使用联合探针锚定连接技术进行大规模人类全基因组测序,2009年报道了其第一例人类全基因组测序并开始提供商业服务。2007年454 Life Sciences创始人Jonathan Rothberg成立了Ion Torrent公司,使用半导体芯片通过检测pH变化进行高通量序列快速测定,2010年其测序仪Ion Proton正式上市。2013年11月,Illumina公司的MiSeqDx Platform获FDA批准。2014年6月30日,国家食品药品监督管理总局(简称CFDA)经审查,批准了华大基因的BGISEQ-1000基因测序仪、BGISEQ-100基因测序仪和胎儿染色体非整倍体(T21、T18、T13)检测试剂盒(联合探针锚定连接测序法)、胎儿染色体非整倍体(T21、T18、T13)检测试剂盒(半导体测序法)的医疗器械注册,这是CFDA首次批准注册的第二代基因测序诊断产品,标志着第二代基因测序产品真正得到国内认可并进入了临床使用。经过十多年发展,第二代测序技术已经实现了千(美)元基因组测序,时间上能缩短至十几天甚至几天。快速发展的高通量测序技术已经被广泛应用于科学研究中,并将逐步应用于疾病的临床检测。
以2009年HeliScope公司和2010年美国太平洋公司开发的PacBio RS测序平台为代表的测序仪被称为第三代测序仪。第三代测序仪与第二代测序仪相比,从技术上实现了测序前文库制备过程中免除PCR放大信号的步骤,即实现了单分子测序。这种革新不仅缩短了整个测序前文库制备时间,也减少了由于PCR过程引入的系统误差,从根本上保证了测序结果的准确性。
在第三代测序平台发展得如火如荼的时候,第四代测序仪在飞速地发展(图18-1),引起了科学家们和临床工作者们的关注。第四代测序平台大多为纳米孔测序仪,通过测定不同碱基通过纳米孔时所引起 的电流变化解读DNA序列。目前英国Oxford Nanopore公司、美国Genia公司、Stratos公司等都在开发针对纳米孔测序技术的相关平台,其中Genia公司已于2014年6月被Roche公司收购。
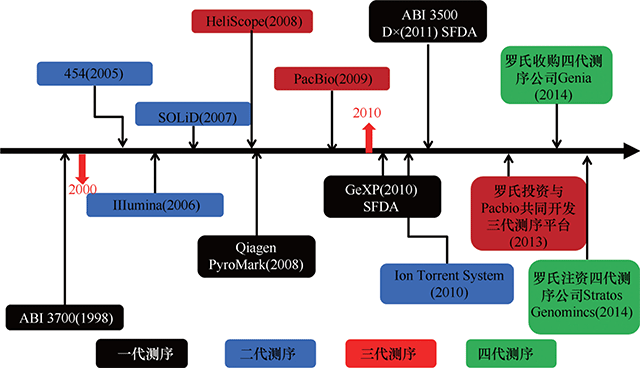
图 18-1 测序技术发展史
1.第一代DNA测序技术 1977年,Sanger等提出了经典的双脱氧核苷酸末端终止测序法。该方法的原理是通过ddNTP随机中断合成待测序列。此后,在Sanger法的基础上,20世纪80年代中期出现了以荧光标记代替放射性同位素标记、以荧光信号接收器和计算机信号分析系统代替放射性自显影的自动测序仪。另外,90年代中期出现的毛细管电泳技术使得测序的通量大为提高。美国应用生物系统公司的3500xL Dx系列产品就是基于第一代DNA测序技术24道毛细管的自动化Sanger测序仪,应用于多种基因测序。
2.第二代DNA测序技术 经过不断开发和测试,进入21世纪后,以Roche公司的GX FLX +技术、Illumina公司的Solexa技术和ABI公司的SOLiD技术和CG公司的LFR技术为标志的第二代测序技术诞生了。与第一代技术相比,第二代测序技术不仅保持了高准确度,而且大大降低了测序成本并极大地提高了测序速度。第二代测序技术最显著的特征是高通量,一次能对几十万到几百万条DNA分子进行序列测序,使得对一个物种的转录组测序或基因组深度测序变得方便易行。
3.第三代DNA测序技术 如果说第二代测序技术的显著特征是高通量,第三代测序技术的显著特征则是长读长。以美国太平洋公司的PacBio RSⅡ机器为代表,在保持高准确度的同时可明显将测序读长再提高10~50倍。第三代测序技术解决了第二代测序技术在测序文库制备时所必需的PCR放大过程,一定程度上消除了PCR所引入的系统误差,同时也减少了整体实验运行时间。
4.第四代DNA测序技术 第四代DNA测序技术同样属于单分子测序,但其利用纳米孔芯片检测单分子测序信号的技术原理不再依赖高速摄像机或者高分辨率的CCD相机,最大程度上降低了检测设备的成本,由于与第三代测序技术相比仍有质的飞跃,通常称之为第四代测序技术。以Roche收购的Genia公司为主要代表的第四代测序技术,能够在提高测序读长的基础上进一步降低测序成本,能为科研、临床诊断提供更灵活快捷的技术平台。
针对基因测序仪的设计、制造,国家质量监督检验检疫总局、国家标准化管理委员会于2014年7月24日发布2014年第19号公告,包括《高通量基因测序技术规程》,标准编号为GB/T 30989-2014,实施日期为2015年3月1日。
针对基因测序仪结果的质量控制,2014年10月,国家标准化管理委员会下达的2014年第一批国家标准制修订计划中包括《高通量基因测序结果评价要求》,该计划编号为20140300-T-469。起草单位包括深圳华大基因研究院、广东省标准化研究院、广东产品质量监督检验研究院、深圳华大基因科技有限公司。
1.基因测序技术在感染性疾病诊断上的应用 基因序列分析目前是分子诊断的金标准,广泛应用于感染性疾病的诊断。测序技术能精确分析碱基序列,通过序列比对,可对各种病原体直接进行鉴定、分型和溯源。测序技术可以用于新发现病原体如新流感病毒、新冠状病毒的鉴定,进行全基因序列分析比对;部分片段测序用于分子分型如针对细菌16SrRNA、真菌18SrDNA、流感病毒HA / NA、肠道病毒71型VPI区的测序;多位点序列分型已成为国际间菌株比较的常用工具,其通过测定6~10个管家基因序列,确定序列型,从而分析菌株间的相互关系。该方法具有分型能力强、结果明确和易于标准化的优点。高通量测序技术是对传统测序的一次革命性改变,一次可以对几十万甚至几百万条DNA分子进行序列分析,同时满足临床病原学检验对时间和准确性上的需求。一项发表在PLoSONE的研究通过对11例极低出生体重婴儿的肠道样本进行测序,表明出生体重低的婴儿肠道微生物群落中存在各种各样的细菌、真菌和蛔虫,其中包括可能的病原体。此次研究突显了出生后肠道内真菌多样性的情况,并提供了存在蛔虫、人类和细菌病毒等许多潜在的病原细菌的证据,进一步的研究将确定这些微生物的来源,以及它们如何影响早产儿的健康,包括侵入性感染,坏死性小肠结肠炎,对成长、神经发育及免疫系统的影响。高通量测序技术有着传统测序技术不可比拟的优势,可以进行大规模平行测序,灵敏度高且单个碱基测序成本低廉。
2.基因测序技术在遗传性疾病诊断上的应用 单基因遗传病是指受一对等位基因控制的遗传病,有7000多种,并且每年在以10~50种的速度递增。也许有人认为单基因遗传病距离自己很遥远,因为单基因遗传病单个发病率低,所以缺乏关注、缺乏治疗手段、缺乏保障体系。单基因遗传病是由基因突变而导致的疾病,具有遗传性和终身性的特点,一旦患上,终身无法治愈。单基因遗传疾病单个发病率低,但种类极多,所以总体发病率高,除部分单基因遗传病可通过手术校正外,大部分往往致死、致残或致畸。较常见的有红绿色盲、血友病、白血病等。单基因遗传病涉及多个学科,临床症状复杂,诊断较为困难,传统检测技术存在漏诊、误诊的风险,可能导致患者错过最佳治疗时机。基因检测能够实现单基因遗传病的早发现、早治疗。有家族遗传史的人群通过基因检测可为婚育提供指导,所以建议通过婚前、孕前和产前的基因筛查来进行预防。
国内单基因遗传病基因检测技术主要采用多重连接探针扩增技术和第一代测序技术,只能对少数几种单基因遗传病进行检测,存在漏检率高、检测通量低的缺点。利用高通量测序技术结合序列捕获技术,则可以覆盖多至上百种单基因遗传病的同时检测,为临床诊断和突变筛查提供参考。其基本策略为利用已知单基因遗传病与突变位点的关系,针对这些区域设计序列捕获探针序列。从被检测人员身上提取血液样本,从中提取基因组DNA,利用NimbleGen序列捕获SeqCap技术,对设计捕获的区域捕获后进行高通量测序,直接检测基因组中DNA序列,通过准确的深度测序结果,查看等位基因突变情况,判断是否存在单基因遗传病,或者根据孟德尔定律,判断后代患病概率。
在以往对多基因遗传病的发病机制探索中,研究者们通过疾病与正常对照样本的基于SNP芯片的全基因组GWAS实验,寻找到了两组个体中多态性与该遗传病的相关性。然而许多疾病的发病又包含后天的因素,如糖尿病与饮食即后天的生活环境有很大的关联,这类复杂的疾病遗传机制很难通过基于SNP芯片的GWAS找到全部决定性的突变。随着二代测序技术及序列富集技术的改进,人们将解决这个问题的注意力转向了与疾病相关的基因编码区域。
全基因组外显子测序是利用序列捕获技术将全基因组外显子区域DNA捕捉并富集后进行高通量测序的基因组分析方法。由于其具有对常见和罕见变异高灵敏度,能发现外显子区绝大部分疾病相关变异及仅需要对约1%的基因组进行测序等优点,促使全基因组外显子测序和基于其发展起来的靶向捕获技术成为鉴定孟德尔疾病致病基因最有效的策略,也被运用于复杂疾病易感基因的研究和临床研究中。在由安徽医科大学第一附属医院牵头的一个银屑病遗传机制研究项目中,就通过靶向富集深度测序获得致病基因在后续群体的验证中获得了更多的阳性结果。对于遗传性疾病,尤其是多基因遗传疾病的致病机制研究,这条崭新的技术研究路线即全外显子组和靶向区域富集的小范围研究,在不会产生大量测序费用的同时可加深测序深度,集中在功能基因的编码区域寻找与疾病相关的突变位点,是对之前基于SNP芯片的全基因组关联研究(GWAS)的一个强有力的补充,同时为未来临床诊断提供更多的生物标记。
3.基因测序技术在肿瘤诊断上的应用 单独遗传因素造成肿瘤的概率<5%。肿瘤的发生主要是遗传基因和环境因素共同作用的结果,其中遗传基因是内因,与人体是否具有肿瘤易感基因有关。肿瘤易感基因检测就是针对人体内与肿瘤发生、发展密切相关的易感基因进行的,它可以检测出人体内是否存在肿瘤易感基因或家族聚集性的致癌因素,根据个人情况给出个性化的指导方案。肿瘤易感基因检测特别适合家族中有癌症病例的人群,可以帮助这类人群提前了解自身是否存在肿瘤易感基因。已知的肿瘤易感性基因有Rb1、WT1、APC、BRCA1、hTERC和Ras等,分别与视网膜母细胞瘤、Wilms瘤、家族性腺瘤性息肉( FAP)、乳腺癌、宫颈癌、消化道肿瘤、泌尿系肿瘤、血液肿瘤等相关。采用DNA直接测序、荧光定量PCR、免疫组化技术等检测相应基因的改变,可评价个体患病风险,进行疾病的早期预防和诊断。
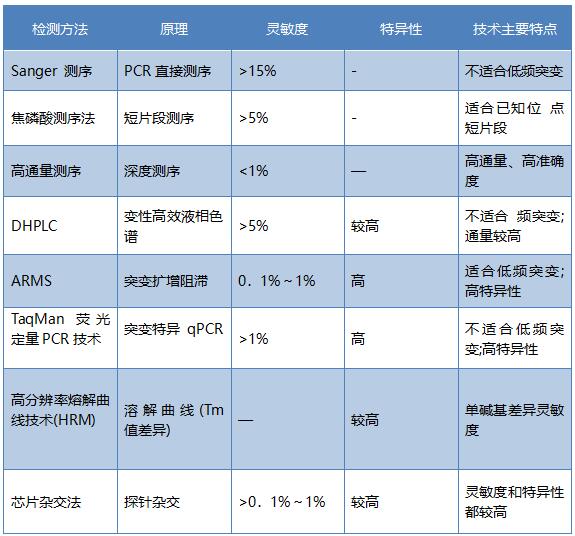
新的分子诊断技术不断出现,同时不同技术方法的组合或联合大大提高了肿瘤分子诊断水平,包括诊断的特异性、敏感性和准确性。序列捕获技术和新一代高通量测序技术相结合也能很好地应用在肿瘤标志物诊断方面,如针对BRCA基因,携带BRCA突变的乳腺癌患者,在初次诊断为乳腺癌之后的10年内,有40%发展出新的原发性乳腺癌,有6%~12%发展出卵巢癌。携带BRCA突变的卵巢癌患者,同时可能患有腹膜癌、输卵管癌,这种癌症在后期难以鉴别。BRCA-1突变携带者倾向于早发型卵巢癌,BRCA-2突变携带者在绝经期后发病率显著提高。可通过Multiplicon CE/IVD BRCAMASTR v2.1或Fluidigm Access Array BRCA1 Kits先对病患的BRCA-1基因外显子区域进行捕获和PCR反应,然后加入区分样本的标签序列后使用454进行测序。不仅提高了同时检测样本的通量,也提高了突变频率的检测灵敏度,便于医生在第一时间掌控病患的情况。基于肿瘤DNA突变检测技术及特点见表18-1。
表 18-1 基于肿瘤DNA突变的检测技术及特点
4.基因测序技术在药物易感基因上的应用 药物研究结果表明,一些基因变异可以使得不同个体对药物产生不同反应。相同的药物,可能在不同的个体中引起包括严重副作用、正常作用、完全无效等不同的用药结果,其原因之一在于个体的某些基因基因型的差异。因而,在床旁诊断中包括药物基因检测结果,对于减小副作用、最大化药效、合理选择药物非常重要。而目前这一实践受到限制的原因在于传统药物基因组检测,一般仅在用药前进行基因检测,影响用药的及时性、延误治疗时机;而普通电子医疗记录尚未能包含药物基因检测结果、基因与用药关系的信息,也影响了个性化治疗的开展;另外,对于根据药物基因组检测结果指导用药是否能提高经济效益及临床结果也尚缺乏大样本数据支持。
在基因突变检测中,除CYP2D6基因外,其他药物基因变异均可通过定向二代测序方法完成:首先采集病人血样、提取基因组DNA,再用序列捕获探针富集样本中药物易感基因的DNA片段,而后进行二代测序。这一方法可以帮助同步完成数十病人、大量基因的同步测序,时间成本及检测费用都相对较少。目前,美国梅奥诊所通过统计学模型,根据年龄、性别、种族和6种慢性病记录,从病人数据库中选出了在3年内有较高可能性会接受斯达汀类药物进行治疗的病人,向这些病人发出邀请。邀请他们自愿参与一组药物基因的变异检测,检测结果将记录在他们的电子医疗档案中,这些基因变异检测的结果可供一些药物使用时参考。此次挑选进行捕获测序的基因包括84个已知与用药相关的基因,由美国药物基因组研究网络(the Pharmacogenomics Research Network,PRN)开发,检测结果分别用于临床相关的用药指导和研究。基因测序结果进行分析后,根据现有的基因与药物关系信息生成用药指导信息,保存在电子档案中。值得一提的是,其他的测序结果也被保留下来,如在将来获得新的基因变异与用药关系的证据,可以重新对基因结果再次进行分析,并将新的用药指导信息添加到电子档案之中。事实证明,这项技术可用于评估提前进行药物基因检测及利用电子医疗档案中基因信息指导用药的作用,可能开创出新的利用基因检测结果指导用药的方法。
5.基因测序技术在无创产前筛查上的应用 人类绝大多数的DNA位于细胞中,但不管是健康的人群还是患病人群,都有小部分DNA位于细胞外,称为游离DNA(cell-free DNA)。外周血中也存在游离DNA,称为外周血循环DNA(circulatingDNAin plasma or serum)。香港科学家卢煜明(音译)1997年发现孕妇血液中存在胎儿DNA,可通过对胎儿DNA进行高通量测序或芯片技术进行分析,筛查其患21染色体三体、18染色体三体、13染色体三体等综合征的风险,被称为NIPS(non-invasive prenatal screen)无创产前筛查或 NIPT(non-invasive prenatal testing)无创产前检测。
我们假定母体外周血中1000基因组拷贝/ml血浆,大部分是母体的,95%来源于母体,5%是胚胎的,1000个基因组中有950个是母体基因组,有50个胚胎基因组,对于母体来说,21号染色体它的基因组含量,对于母体二倍体都是1900∶1900,对于胎儿它的基因组拷贝是100∶100,对于三倍体是100∶150,总的来说母体外周血浆对于二倍体来说是2000∶2000,对于三倍体来说有轻微的增加就是2000∶2050,这么小的一个变异需要一个高分辨率的方法才能检测出。也就是检测出2.5%胚胎DNA含量,相当于胚胎基因组比例5%的一半,就是2.5% ,二代测序技术是完全可以做到的。
目前,国外有四家公司提供无创产前筛查,它们分别是Ariosa Diagnostics(已被Roche收购)、Natera、Sequenom和Verinata Health(已被Illumina收购)。该检测通常是通过高通量测序技术和DNA芯片技术筛查孕妇血液中的胎儿DNA,以了解13、18和21号染色体的非整倍体风险,并开始筛查性染色体的非整倍体。
传统的产前筛查技术如血清学筛查和超声检查,虽无风险,但检出率不高,大于35岁的产妇生出唐氏综合征婴儿的风险率为1/260,因而不适宜接受唐筛及超声检查,而是直接要求接受羊水穿刺这类产前诊断。然而,这一用一根长长的针扎破羊膜的检查虽可100%确诊,却有0.5%左右的流产率。无创产前筛查技术的革新能为这些风险较高,如年龄较大、有家族史,或者超声波检测异常的孕妇带来无风险的便利检测,提高检出率,降低流产风险,成为产前筛查技术未来的重要发展方向。
(一)临床实验室相关管理规范
临床基因测序实验室的设置和管理需遵从《医疗机构临床基因扩增检验实验室管理办法》(卫办医政发〔2010〕194号),详情参考“第十七章核酸提取仪和PCR仪”的质量管理部分。
按照2014年02月17日国家食品药品监督管理总局、国家卫计委办公厅下发的《关于加强临床使用基因测序相关产品和技术管理的通知》,通知明确指出,基因测序产品需经食品药品监管部门审批注册,并经卫生计生行政部门批准技术准入方可应用。也就是说,基因测序临床应用所用仪器归口CFDA管理,临床使用归口卫计委或属地管理。
(二) 测序仪管理要求与程序
以下以ABI3130为例,描述测序仪日常维护与保养的基本步骤:
(1)相关耗材的使用
1) Hi-Di:分装以避免反复冻融。
2) 胶:4℃保存,用之前2h从冰箱中取出回温,并且不同种类的胶不能混用:310pop4,310pop6,3100pop4、pop6、pop7,更换胶时请使用相关的wizard。
3) 水:使用电导率为18.2MΩ的水。
4) Buffer:新鲜配制。
5) 毛细管:未使用过的毛细管请置于干燥的地方保存;用过的毛细管两端一定不能离开水。
(2) Spatial校正。
(3) Spectral校正。
(4) 每天要做的事情
1) 运行前检查block有无气泡。
2) 检查Buffer及水的高度。
3) 注意机器的环境卫生。
4) 检查激光风扇出口是否被堵住。
(5 ) 每两天要做的事情:更换水、Buffer,且Buffer要新鲜配制。
(6) 每周要做的事情
1) 更换新鲜的胶。
2) 清洗大小注射器。
3) 更换3130测序仪PDP water seal trap中的水。
4) 重新启动电脑和测序仪。
(7) 每月要做的事情
1) 清洗上下胶引导块。
2) 清洗毛细管的扫描窗口并做spatial。
3) 对于3130,清洗pump block,请使用waterwash wizard。
4) 对电脑进行碎片整理并备份相关数据。
5 ) 数据库整理:cleanupDB.bat,cleanup processed plate。
(8) 每半年或一年要做的事情
1) 更换毛细管或根据需要更换。
2) 更换注射器。
3) 根据需要(如spatial信号变低等)可联系工程师对机器进行保养。
 京公网安备11010102003307号
京公网安备11010102003307号

微信扫一扫
关注医隆网